iRepertoire 的数据分析服务
我们的所有服务均通过专有流程提供免费的基本数据分析,然后将数据输出到iRweb。iRweb是一款基于网络的RepSeq生物信息学软件,允许研究人员对其样本执行以下所有服务和分析:
- 条形码解复用
- 库中捕获的 CDR3 数量以及每个样本中唯一 CDR3 的数量
- 每个样本中免疫组库的多样性,通过多样性指数、专有 D50 值和香农熵来捕获
- V/J 使用、V/J 修剪、CDR3 长度和 N 添加的标准化和非标准化分布。(标准化通过将每个独特的 CDR3-VDJ 组合视为一个而不管读取数如何来解释不同的覆盖深度。)
- VDJ-C映射
- 与 IMGT 数据库比对
- VJ 组合分布为二维和三维地图
- CDR3 分级肽频率映射,包括 BCR 类别转换
- CDR3 肽与 VJ 频率列表
- CDR3 代数:对不同的样本进行缩放,以便可以跨具有不同读取深度的样本比较 CDR3 的频率。
购买了我们的试剂盒的客户也可以访问 iRweb 和进行基本数据分析,但为了避免费用,需遵守某些规则和限制。
通过我们的生物信息学页面了解有关 iRweb 的更多信息并探索演示。
报告
可以通过 iRweb 以各种报告(如热图、树状图以及多样性的图形和数字表示)对数据进行可视化。数据以预渲染的 PDF 或 JPEG 文件形式提供,但如果研究人员想要自己渲染图形,也可以以 CSV 形式提供。
热图
热图显示群体内不同等位基因的相对频率。例如,您可以使用热图来直观地了解健康患者和癌症患者中哪些 T 细胞受体基因变体出现频率最高。多样性(不同变体的数量)的定量差异在 2D 或 3D 热图中一目了然,如下图所示。

树形图
多样性也可以使用树状图来显示。下图中的树状图表示变异种群,其中每个克隆型由不同颜色的形状表示。形状的大小反映了变异的频率(即,大形状代表高频率或克隆扩增)。树状图可以快速直观地评估不同样本中的相对库多样性;形状越小、颜色越多样,多样性就越大。

图表
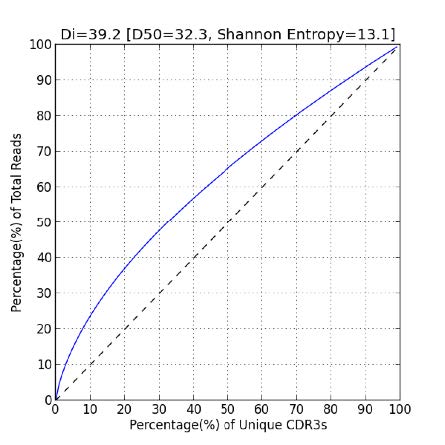
免疫库的多样性也可以通过多样性指数 (Di)、香农熵和 D50 值以数字形式捕获。多样性指数是每个 CDR3 的频率和唯一 CDR3 总数的函数。香农熵仅涉及前 10,000 个最常见的 CDR3。D50 是占样本中 CDR3 总数 50% 的显性克隆和唯一克隆的百分比。非常多样化的库的 D50 值将接近 50。多样性由下图表示,其中黑色虚线代表“完美”多样性。
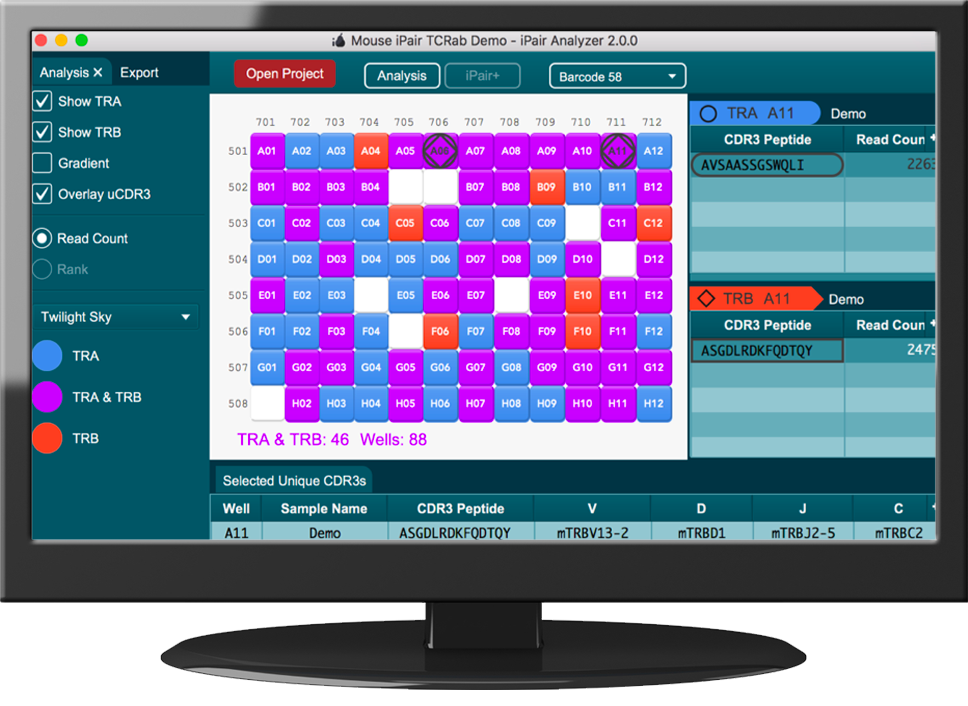
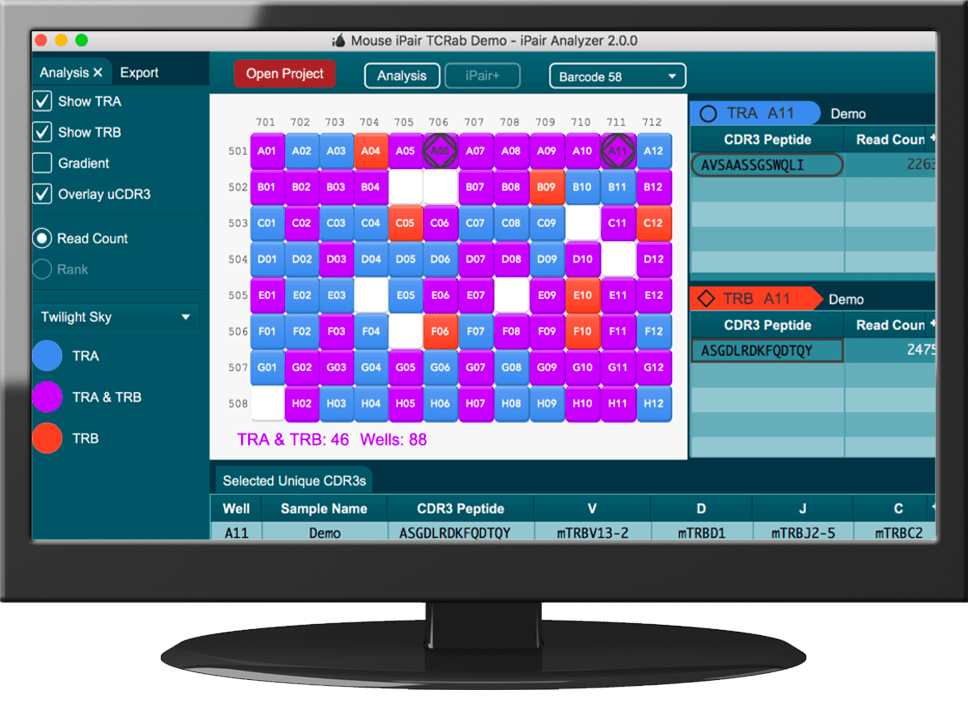
iPair 分析仪
iPair Analyzer是专为通过我们的 iPair 服务执行的单细胞组测序数据而开发的数据分析平台。iPair Analyzer 有助于可视化和比较单细胞以及批量组的数据(如果批量测序是串联进行的)。
iPair Analyzer 独有的图形用户界面在交互式面板中显示 96 孔板中的样本。面板上可通过不同颜色和符号突出显示读取深度、链类型或特定独特 CDR3 的存在等特征。选择面板中的单元格时,将显示单细胞 BCR 或 TCR 链结果。工作表功能可让您选择感兴趣的链,并进一步探索和比较其他特征,例如读取的核苷酸序列、CDR1、CDR2、CDR3、V、D 和 J 使用情况。